Open: Обработка конформационных ансамблей в Python: Difference between revisions
mNo edit summary |
|||
| Line 202: | Line 202: | ||
</source> | </source> | ||
В результате мы получили DataFrame, в котором MyCondition соответствует тому, выполняются наши требования на | В результате мы получили DataFrame, в котором MyCondition соответствует тому, выполняются наши требования на водородные связи или нет: | ||
MyCondition Energy | MyCondition Energy | ||
Revision as of 12:11, 11 September 2022
В чем заключается эта обработка и для чего её делать?
Программы для конформационного поиска обычно выдают результаты в виде XYZ файлов, структуры из которых обычно надо вписать в инпут-файлы для последующей квантовохимической оптимизации. Чтобы уменьшить количество расчетов вам может захотеться сделать следующие операции, перед тем, как генерировать инпут-файлы:
- Отсеять конформации с чрезмерно высокой энергией;
- Удалить дубликаты;
- Проверить сохранение констрейнов и отбросить структуры, в которых они нарушены. Например, когда вы ищите конформеры переходного состояния в CREST, полученный конформационный набор может содержать структуры, в которых заданные констрейны сильно нарушены. Если начнете оптимизировать такие геометрии в DFT, то ПС не найдется, а вы потратите время.
- Проанализировать ансамбль и узнать, какие слабые взаимодействия (водородные связи и т.п.) могут образовываться в вашей молекуле. Найденные контакты можно учесть в дополнительных запусках конформационного поиска с констрейнами, чтобы лучше перебрать варианты с данными взаимодействиями.
- Объединить конформационные ансамбли из нескольких расчетов, полученные, например, как в пункте выше, и отсортировать структуры по их энергии.
Этот мануал посвящен моей библиотеке pyxyz, которая позволяет решать эти задачи несколькими строчками в Python. Также этот мануал может быть полезен при анализе XYZ-файлов, полученных в результате расчета молекулярной динамики, оптимизаций, сканов, IRC и т.п.
Установка библиотеки
Для работы под Linux требуется Python версии 3.7 или выше. Для работы под Windows требуется Python версии 3.8 или выше. Из библиотек для Python будет нужен только Numpy.
Для установки на Linux нужно выполнить в терминале следующую команду:
pip install -i https://test.pypi.org/simple/ "pyxyz<1.0"
Для установки на Windows нужно выполнить в командной строке эту строчку:
pip install -i https://test.pypi.org/simple/ "pyxyz>=1.0"
Обратите внимание, что в некоторых случаях может понадобиться заменить pip на pip3 (это зависит от того, как у вас установлен Python).
После вызова pip install проверьте корректность установки: импортируется ли библиотека и выполняются ли тесты:
import pyxyz
import pyxyz.test
pyxyz.test.run_tests()
При импорте pyxyz под Linux может возникнуть ImportError, который исправляется изменение переменной LD_LIBRARY_PATH. Как это исправить библиотека пишет во время загрузки (строчка начинается с [WARNING]).
Ошибка выглядит так:
>>> import pyxyz
[WARNING] In case you don't have gslcblas installed and get ImportError, call 'export LD_LIBRARY_PATH=/my/path/to/pyxyz:$LD_LIBRARY_PATH'
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
File "/home/xray/.local/lib/python3.8/site-packages/pyxyz/__init__.py", line 10, in <module>
from confpool import Confpool
ImportError: libgslcblas.so.0: cannot open shared object file: No such file or directory
То есть перед запуском python нужно выполнить эту строчку и ImportError исчезнет (/my/path/to/pyxyz надо заменить на то, что будет у вас):
export LD_LIBRARY_PATH=/my/path/to/pyxyz:$LD_LIBRARY_PATH
Пример обработки набора конформаций
Архив с примерами -- Pyxyz_manual.zip (File:Pyxyz manual.zip)
В качестве примера рассмотрим молекулу из файла diol_single.xyz и её конфнабор из CREST - diol_crest.xyz (см. архив). Рассмотрим несколько учебных задач, которые могли бы возникнуть на практике. Обратите внимание, что в пределах одного объекта Confpool количество атомов в анализируемых молекулах и последовательность атомов в импортируемых xyz-файлах должны быть постоянными.
Для работы потребуются следующие импорты:
from pyxyz import Confpool
import numpy as np
Для большей интерактивности с библиотекой можно работать не только запуская код из файла, но и через jupyter notebook.
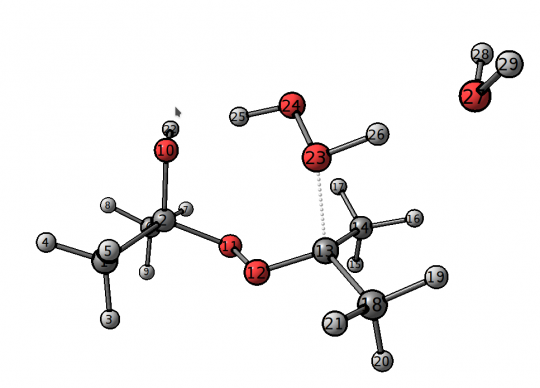
Базовая обработка конформационных наборов
Допустим, мы сделали два запуска CREST и получили два файла с конформациями crest_conformersA.xyz и crest_conformersB.xyz. Для начала, загружаем оба xyz-файла в один объект Confpool:
p = Confpool()
p.include_from_file("crest_conformersA.xyz")
p.include_from_file("crest_conformersB.xyz")
print("Loaded {} conformers".format(p.size))
Для дальнейшей обработки кроме геометрий нам понадобятся энергии конформеров, их можно извлечь из описания к структурам:
H2KC = 627.509474063
KC2H = 1 / H2KC
n_del = 0
p["Energy"] = lambda m: float(m.descr.strip())
n_del += p.upper_cutoff("Energy", 5.0 * KC2H)
print("{} confs were filtered out".format(n_del))
p.sort("Energy") # Ascending by default. Use ascending=False to override
Четвертая строчка здесь самая интересная - в ней мы говорим, что каждой конформации нужно сопоставить ключ Energy и рассчитать его из описания к конформации с помощью переданной функции (strip удаляет ненужные пробелы, а float конвертирует в дробное число). Далее мы применям upper_cutoff, чтобы отбросить все конформеры с энергией выше 5 ккал/моль относительно минимальной энергии в наборе, и сортируем по возрастанию энергии. Стоит отметить, что все операции, сопровождающиеся удалением конформеров из набора (всего их четыре: filter, rmsd_filter, upper_cutoff, lower_cutoff), возвращают целое число - количество удаленных структур. Завершаем обработку отбрасыванием дубликатов по RMSD:
n_del += p.rmsd_filter(0.3)
print("{} confs were filtered in total".format(n_del))
p.descr = lambda m: "Conf #{}; Energy = {:6f} a.u.".format(m.idx + 1, m["Energy"])
p.save('basic_processing.xyz')
Здесь мы передали критерий для фильтрования RMSD < 0.3 A. Затем, для оставшихся уникальных конформеров обновили описание с помощью переданной lambda-функции и сохранили набор в файл. Как альтернатива, можно сразу сгенерировать input-файлы для оптимизации или ещё чего-то:
gjftemplate = """\
%nprocs=24
# opt=(tight,maxcycle=300) freq=noraman def2tzvp empiricaldispersion=gd3bj rpbe1pbe scrf=(cpcm,solvent=water)
Conformation_{idx}
0 1
{xyz}
"""
for i in range(p.size):
xyz = p[i].xyz # xyz is a Numpy matrix
atom_symbols = p.atom_symbols
xyzlines = []
for j, symbol in enumerate(atom_symbols):
xyzlines.append("%2s %14.6f %14.6f %14.6f" % (symbol, *xyz[j]))
with open("conf_{}.gjf".format(i), 'w') as f:
f.write(gjftemplate.format(xyz="\n".join(xyzlines), idx=i))
В 90% случаев, как мне кажется, на этом этапе потребности расчетчика должны уже быть удовлетворены. Но иногда может захотеться обработать структуры более детально.
Анализ геометрии
Продолжаем работу над объектом p, полученным в предыдущей секции. Как видно из рисунка 1, явная молекула воды была добавлена, чтобы стабилизировать атом водорода 26, но в ходе конфпоиска H2O могла перескочить на другие атомы. Чтобы понять, сколько структур содержат интересующую нас водородную связь H26-O27, можно выполнить следующий код:
p["H2O dist"] = lambda m: m.l(26, 27)
for i in range(p.size):
print("Length = {}".format(p[i].l(26, 27)))
# the same as
print("Length = {}".format(p[i]["H2O dist"]))
# Look at the printed numbers. And then
print("Found {} good structures".format(p.count(lambda m: m["H2O dist"] < 1.8)))
Мы здесь использовали функцию m.l(a, b), чтобы обратиться к длинам связи отдельной структуры в наборе (внутри lambda-функции и при обращении по индексу). И ещё пригодилась функция count, чтобы посчитать количество структур, удовлетворяющих заданному условию. Небольшое отступление, удобством в обращении по ключу в цикле, т.е. p[i]["H2O dist"], является возможность поменять значение ключа только для i-ой структуры: p[i]["H2O dist"] = 123.0; но в текущей задаче это не понадобилось.
Далее, можно отобразить наличие нужно водородной связи в описании структуры:
def quality_check(m):
if m["H2O dist"] < 1.8:
comment = "Nice"
else:
comment = "Garbage"
return "{}; E={:3f} a.u.".format(comment, m["Energy"])
# Print new descriptions in the console
for i in range(p.size):
print(quality_check(p[i]))
p.descr = quality_check # Set new descriptions
p.save('check.xyz') # Now show this file to your supervisor
Если нужно наложить какие-то сложные требования на геометрию, то это можно сделать в одну стручку. Валентные и торсионные углы вычисляются функциями m.v(a, b, c) и m.z(a, b, c, d), соответственно:
hbond_condition = lambda m: m.l(25, 10) < 2.0 and m.v(24, 25, 10) > 150.0 and abs(m.z(26, 23, 24, 25)) > 120.0
print("There are {} matches".format(p.count(hbond_condition)))
p.filter(hbond_condition)
min_ener = min(p["Energy"]) # p["Energy"] creates a list of floats
p["E.Rel"] = lambda m: H2KC * (m["Energy"] - min_ener)
p.descr = lambda m: "{}; E(rel.)={:3f} kcal/mol".format(m.descr, m["E.Rel"])
p.save('check.xyz')
Не считая сложного условия hbond_condition, в части с фильтрованием всё очевидно. А вот во второй части кода есть особенности: обращением p["Energy"] можно получать лист значений некого существующего ключа, а при установке новых значений p.descr или p["Some key"] можно обращаться к их старым значениям.
Тонкости работы с библиотекой
Как вы уже могли заметить, библиотека сделана исходя из нескольких ограничений:
- Все структуры подаются с одинаковой нумерацией атомов. Если дать две структуры с нумерациями C1, H2, H3 и H1, C2, H3, то будет исключение.
- Кроме XYZ, каждой структуре сопоставляется одна строка m.descr и сколько угодно дробных чисел m["SomeKey"], к которым можно обращаться по ключу (ключами могут быть только строки). Попытка записать по ключу что-то кроме дробного числа приведет к исключению (аналогично, с нестрочными описаниями структур).
- Все структуры имеют одинаковый набор ключей.
Вернемся к задаче с переходным состоянием с Рисунка 1 и допустим, что мы хотим проанализировать, насколько энергетически выгодны конформации, заданные критерием hbond_condition(m) and m.l(26, 27) < 1.8, на фоне остальных найденных конформаций.
import pandas as pd
p = cp.Confpool()
p.include_from_file(os.path.join(curdir, "testsetA.xyz"))
p.include_from_file(os.path.join(curdir, "testsetB.xyz"))
p["Energy"] = lambda m: float(m.descr.strip())
p.sort("Energy")
p.rmsd_filter(0.3)
for i in range(p.size):
if hbond_condition(p[i]) and p[i].l(26, 27) < 1.8:
# The key "MyCondition" didn't appear above, but it's okay
p[i]["MyCondition"] = 1.0 # p[i]["MyCondition"] = 1 gives exception
else:
p[i]["MyCondition"] = 0.0 # same exception with 0
print(pd.DataFrame(p.as_table()))
В результате мы получили DataFrame, в котором MyCondition соответствует тому, выполняются наши требования на водородные связи или нет:
MyCondition Energy 0 0.0 -45.344433 1 0.0 -45.343703 2 1.0 -45.343607 3 0.0 -45.343443 4 0.0 -45.342388 5 1.0 -45.342137 ...
Здесь, мы пользуемся небольшим костылем в связи с тем, что ключи обязаны быть дробными числами, поэтому MyCondition - дробное число, а не bool, например. Чтобы об этом не забывать, никакие неявные конверсии во время присвоения не делаются (см. выше, что с 1.0 работает, а с 1 - нет). Другой момент - это что присвоение значения нового ключа для одной структуры автоматически инициализирует этот ключ со значением 0.0 для всех остальных структур (см. третий принцип в начале раздела). Ну и, наконец, p.as_table() генерирует вам данные всех ключей в виде словаря из листов.
Подробное описание функционала библиотеки
Описание методов класса Confpool приведено по следующей схеме:
Имя функции(название аргумента: тип аргумента, ...) -> тип возвращаемого значения
Во всех примерах объект Confpool имеет название e. То есть он создавался строчкой:
e = Confpool()
Добавление структур в набор
include_from_file(filename: str, energy=None or function) -> None, energy(descr: str) -> float
Загрузка структур из XYZ-файле filename и (опционально) парсинг энергии из описания каждой структуры, с помощью функции energy.
filename - имя xyz-файла для импорта;
energy - keyword, который можно игнорировать или передавать функцию для выделения энергии из строчки описания в xyz-файле.
Пример, когда в описании к структуре энергия дается, как число без дополнительных подписей:
e.include_from_file("file.xyz", energy=lambda x: float(x.strip()))
Изменение данных
sort() -> None Сортировка ансамбля по возрастанию энергии. Пример: e.sort()
update_description(f: function) -> None, f(e: float, d: str) -> str
Используя функцию f сгенерировать новые описания к структурам из их энергии (float, первый аргумент f) и старого описания (str, второй аргумент f).
Пример: e.update_description(lambda ener, d: "E = {} a.u. (old descr = {})".format(ener, d))
Фильтры:
energy_filter(e_cutoff: float, etype='kcal/mol' or 'hartree'/'a.u.') -> None Отбросить все конформеры, энергия которых выше энергии минимального на e_cutoff. В случае, когда etype=kcal/mol, разницы по энергии переводятся из a.u. в kcal/mol, после чего производится сравнение с e_cutoff. Во всех случаях предполагается, что энергия дана в a.u. Дефолтный etype=kcal/mol. Пример: e.energy_filter(5.0, etype='kcal/mol')
distance_filter(idxA: int, idxB: int, condition: function) -> None, condition(dist: float) -> bool Отбросить все конформеры, расстояние между атомами idxA и idxB которых не удовлетворяет условию condition. Функция condition получает на вход расстояние между атомами и, если она возвращает False, то структура отбрасывается. Пример, в котором отбросятся структуры с расстоянием между атомами 1 и 2 больше, чем 1.7: e.distance_filter(1, 2, lambda x: x < 1.7)
valence_filter(idxA: int, idxB: int, idxC: int, condition: function) -> None, condition(angle: float) -> bool Отбросить все конформеры, валентный угол между атомами idxA, idxB и idxC которых не удовлетворяет условию condition. Функция condition получает на вход угол между атомами в градусах и, если она возвращает False, то структура отбрасывается. Пример, в котором отбросятся структуры в углами между атомами 1, 2 и 3 близкими к развернутому: e.valence_filter(1, 2, 3, lambda a: 180.0 - a < 10.0)
dihedral_filter(idxA: int, idxB: int, idxC: int, idxD: int, condition: function) -> None, condition(angle: float) -> bool Отбросить все конформеры, торсионный угол между атомами idxA, idxB, idxC и idxD которых не удовлетворяет условию condition. Функция condition получает на вход угол между атомами в градусах (между -180 и 180) и, если она возвращает False, то структура отбрасывается. Пример, в котором отбросятся структуры в торсионными углами между атомами 1, 2, 3 и 4 близкими к нулю: e.dihedral_filter(1, 2, 3, 4, lambda a: abs(a) < 10.0)
Получение данных
save(filename: str) -> None
Выписать данный конформационный набор в файл с название filename в формате XYZ. Струтуры будут выписаны в определенном порядке, например, в котором они находились в ранее импортированном файле или порядке после сортировки по энергии. В полученном XYZ файле будут включены описания к каждой структуре.
Пример: e.save("result.xyz")
size() -> int
Возвращает количество структур в данном конформационном наборе.
Пример: print("We have {} structures".format(e.size()))
get_structure(index: int) -> dict = {'energy': float, 'descr': str, 'xyz': np.array}
Возвращает данные о структуре с индексом index. Нумерация структур начинается с нуля, то есть 0 <= index <= e.size() - 1. Данные передаются в виде словаря с ключами 'energy' (энергия), 'descr' (описание) и 'xyz' (numpy матрица N на 3).
Пример: print("The first structure has energy {}".format(e.get_structure(0)['energy']))
get_atom_symbols() -> list of str Возвращает лист из символов атомов.
Подсчет структур:
energy_count(e_cutoff: float, etype='kcal/mol' or 'hartree'/'a.u.') -> int Возвращает количество структур в пределах данного e_cutoff от структуры с минимальной энергией (см. подробности в energy_filter).
distance_count(idxA: int, idxB: int, condition: function) -> None, condition(dist: float) -> bool Возвращает количество структур, удовлетворяющих условию condition на длину связей между атомами idxA и idxB (см. подробности в distance_filter).
valence_count(idxA: int, idxB: int, idxC: int, condition: function) -> None, condition(angle: float) -> bool Возвращает количество структур, удовлетворяющих условию condition на валентный угол между атомами idxA, idxB и idxC (см. подробности в valence_filter).
dihedral_count(idxA: int, idxB: int, idxC: int, idxD: int, condition: function) -> None, condition(angle: float) -> bool Возвращает количество структур, удовлетворяющих условию condition на торсионный угол между атомами idxA, idxB, idxC и idxD (см. подробности в dihedral_filter).
Сборка pyxyz из исходного кода
Этот раздел может быть полезным (1) если вы хотите добавить в pyxyz новую функцию или нашли баг, (2) если вам нужно собрать библиотеку под новую версию Python или под нестандартную операционную систему, (3) хотите научиться делать Python-модули с помощью pybind11/C++.
При загрузке на pyxyz на pypi мной используется немного странное соглашение о версии библиотеки: a.b.c, где а - это 1 (версия под Windows) или 0 (версия под Linux), b - обозначение версии Python (37, 38, 39 или 310), c - собственно версия кода библиотеки. Например, pyxyz версии 1.310.3 собирается для работы под Windows на Python 3.10 (и номер ревизии кода = 3). Когда хотите загрузить на pypi новую ревизию кода, увеличивайте номер ревизии (c) в файле setup.py (директории release_files и setup.py):
version='1.3{ver}.1', # здесь версия равна 1 (правая единица)
Сборка под Linux проверена на GCC 10.3.1, а под Windows - на GCC 12.1.0.
Сборка под Linux
Пререквизиты: make, cmake, g++, gsl, boost, pkg-config. Чтобы собрать библиотеку на одном компьютере без цели её распространения (для выполнения конкретной задачи или в процессе разработки), выполните следующее:
git clone --recurse-submodules https://github.com/nkrivoshchapov/confpool.git
cd confpool
python build_script.py 8 # Восьмерка соответствует x в версии Python3.x (может быть 8, 9 или 10)
cd release
pip install .
Готово!
Если стоит задача загрузить библиотеку на pypi, то понадобится ещё и conda, в которой должны быть созданы отдельные среды с разными версиями Python:
conda create -n py38 python=3.8 -y
conda activate py38
pip install numpy twine
conda deactivate
# Повторить для Python3.x (x = 7, 9, 10)
Таким образом, должно быть создано четыре среды: py37, py38, py39 и py310. Далее скачиваем исходный код:
git clone --recurse-submodules https://github.com/nkrivoshchapov/confpool.git
cd confpool
Затем для каждой версии Python выполняем сборку библиотеки и загружаем соответствующую версию на pypi:
conda activate py38
python build_script.py 8 # Восьмерка соответствует x в версии Python3.x
cd release
python setup.py sdist
twine upload --repository testpypi dist/* # Ввести логин и пароль
conda deactivate
Повторите тоже самое для остальных версий Python.
После запусков twine вам будет выписываться ссылка типа https://test.pypi.org/project/pyxyz/0.310.3/, по которой можно будет посмотреть команду для скачивания только что собранной версии. Более того, по аналогии с
pip install -i https://test.pypi.org/simple/ "pyxyz<1.0"
можно написать универсальную команду, чтобы устанавливать независимо от версии Python.
Сборка под Windows
Установите себе MSYS2 по ссылке (там надо нажать на 32-bit или 64-bit, а потом скачать свежий exe-шник msys2-x86_64-???.exe). После установки запустите через Пуск терминал MSYS2 MinGW x64 и установите некоторые пререквизиты:
pacman -Syu
pacman -S --needed base-devel mingw-w64-x86_64-toolchain
pacman -S mingw-w64-x86_64-gsl mingw-w64-x86_64-boost mingw-w64-x86_64-cmake mingw-w64-x86_64-pybind11 git
Стоит отметить, что MSYS2 и установленные только что библиотеки нужны только для сборки pyxyz под Windows, но не требуются для его использования.
Также нужно, чтобы у вас была установлена Anaconda (или Miniconda). Узнайте расположение файла conda.exe и добавьте в файл C:\msys64\home\*myusername*\.bash_profile следующую строчку:
eval "$('/c/tools/Anaconda3/Scripts/conda.exe' 'shell.bash' 'hook')"
# Проверьте путь к conda.exe!
Как и в Linux может быть два варианта сборки: только для своего компьютера и для загрузки на pypi. Инструкции для своего компьютера:
git clone --recurse-submodules https://github.com/nkrivoshchapov/confpool.git
cd confpool
python winbuild_script.py 8 # Восьмерка соответствует x в версии Python3.x (может быть 8, 9 или 10)
cd release
pip install .
Готово! Теперь даже из командной строки Anaconda (вне MSYS2) можно будет вызывать pyxyz.
Как и c линуксом, если стоит задача загрузить библиотеку на pypi, то понадобится создать отдельные среды в conda с разными версиями Python:
conda create -n py38 python=3.8 -y
conda activate py38
pip install numpy twine
conda deactivate
# Повторить для Python3.x (x = 9, 10)
Таким образом, должно быть создано три среды: py38, py39 и py310. Далее скачиваем исходный код:
git clone --recurse-submodules https://github.com/nkrivoshchapov/confpool.git
cd confpool
Затем для каждой версии Python выполняем сборку библиотеки и загружаем соответствующую версию на pypi. Шаги по загрузке на pypi делаются не в MSYS2, а в обычной командной строке Anaconda, потому что MSYS2 зависает на стадии ввода пароля при загрузке через twine.
conda activate py38
python winbuild_script.py 8 # Восьмерка соответствует x в версии Python3.x
conda deactivate
# В отдельном окне командной строки Anaconda (уже не MSYS2):
conda activate py38
cd *mypath*/confpool/release
python setup.py sdist
twine upload --repository testpypi dist/* # Ввести логин и пароль
conda deactivate
Повторяем эти шаги для версий Python 3.9 и 3.10. Универсальная ссылка на скачивание под Windows делается по следующему принципу:
pip install -i https://test.pypi.org/simple/ "pyxyz>=1.0"
TODO
1. RMSD filtering
2. Build connectivity graphs & search for VdW contacts
3. Complex filtering conditions (how to implement?)
4. Complex description templates (how to implement?)
TODO: include_from_dict
TODO: copy constructor, operator+