CCC:HomeWork2: Difference between revisions
m (Если в логе CREST присутствуют проблемы с топологией, то есть screst_noreftopo, он запустит crest в очередь с флагом --noreftopo.) |
m (xtb-control.txt) |
||
| Line 25: | Line 25: | ||
# Для начала нужно нарисовать структуру в [https://www.chemcraftprog.com ChemCraft]. В переходном состоянии все показанные палочками длины связей должны быть близки к равновесным значениям (их можно посмотреть [https://theorchem.ru/mediawiki/images/6/60/OrganicBondLenghts.pdf вот тут]), а разрывающиеся/замыкающиеся связи C...C и C...Cl имеют длины 2.0 и 2.3 ангстрем, соответственно. Также стоит обратить внимание на формы атомов (так, sp3-углероды должны быть тетраэдрическими) и торсионные углы: как правило, заторможенные конформации предпочтительнее заслонённых. | # Для начала нужно нарисовать структуру в [https://www.chemcraftprog.com ChemCraft]. В переходном состоянии все показанные палочками длины связей должны быть близки к равновесным значениям (их можно посмотреть [https://theorchem.ru/mediawiki/images/6/60/OrganicBondLenghts.pdf вот тут]), а разрывающиеся/замыкающиеся связи C...C и C...Cl имеют длины 2.0 и 2.3 ангстрем, соответственно. Также стоит обратить внимание на формы атомов (так, sp3-углероды должны быть тетраэдрическими) и торсионные углы: как правило, заторможенные конформации предпочтительнее заслонённых. | ||
#* Чтобы научиться им пользоваться, выполните все задачи из [https://github.com/medvedev-m/CompChemEdu/blob/master/%D0%A0%D1%83%D0%BA%D0%BE%D0%B2%D0%BE%D0%B4%D1%81%D1%82%D0%B2%D0%BE%20%D0%BF%D0%BE%20ChemCraft.pdf этого мануала]. | #* Чтобы научиться им пользоваться, выполните все задачи из [https://github.com/medvedev-m/CompChemEdu/blob/master/%D0%A0%D1%83%D0%BA%D0%BE%D0%B2%D0%BE%D0%B4%D1%81%D1%82%D0%B2%D0%BE%20%D0%BF%D0%BE%20ChemCraft.pdf этого мануала]. | ||
# Следующая задача: проверить корректность построенной молекулы и выбранных параметров (force constant) перед конф. поиском путём запуска оптимизации в XTB. Запуск XTB требует двух файлов, XYZ и xtb-control (они могут называться и иначе, но файл XYZ обязательно должен иметь именно такое расширение). Запуск XTB: <code>sxtb input.xyz xtb-control</code> <br />xtb-control - это обычный текстовый файл, содержащий:<syntaxhighlight lang="aconf"> | # Следующая задача: проверить корректность построенной молекулы и выбранных параметров (force constant) перед конф. поиском путём запуска оптимизации в XTB. Запуск XTB требует двух файлов, XYZ и xtb-control.txt (они могут называться и иначе, но файл XYZ обязательно должен иметь именно такое расширение). Запуск XTB: <code>sxtb input.xyz xtb-control.txt</code> <br />xtb-control.txt - это обычный текстовый файл, содержащий:<syntaxhighlight lang="aconf"> | ||
$chrg -1 | $chrg -1 | ||
$constrain | $constrain | ||
| Line 46: | Line 46: | ||
</syntaxhighlight>В XYZ-файле в строке 1 идёт число атомов в первой молекуле, далее в строке 2 - комментарий, содержащий любые символы, кроме переноса строки, затем следуют строчки, содержащие название элементов и их координаты X, Y и Z. После завершения первой молекулы (считается из количества атомов в ней), идёт вторая молекула, и так далее - сколько угодно молекул в одном XYZ-файле. | </syntaxhighlight>В XYZ-файле в строке 1 идёт число атомов в первой молекуле, далее в строке 2 - комментарий, содержащий любые символы, кроме переноса строки, затем следуют строчки, содержащие название элементов и их координаты X, Y и Z. После завершения первой молекулы (считается из количества атомов в ней), идёт вторая молекула, и так далее - сколько угодно молекул в одном XYZ-файле. | ||
# После запуска расчёта в XTB, ждём его окончания, переименовываем полученный xtbopt.log в xtbopt_trj.xyz, скачиваем себе на компьютер, открываем в ChemCraft и смотрим, как шла оптимизация. | # После запуска расчёта в XTB, ждём его окончания, переименовываем полученный xtbopt.log в xtbopt_trj.xyz, скачиваем себе на компьютер, открываем в ChemCraft и смотрим, как шла оптимизация. | ||
# Если в визуализации в Chemcraft видно, что оптимизация прошла корректно, констрейны сработали, а молекула ни во что не изомеризовалась (ковалентные связи те же, что и на схеме выше), значит файлы XYZ и xtb-control получились корректными, и можно запускать конформационный поиск в программе CREST: <code>screst input.xyz xtb-control.</code> Если в логе CREST присутствуют проблемы с топологией, то есть <code>screst_noreftopo</code>, он запустит crest в очередь с флагом <code>--noreftopo</code>. | # Если в визуализации в Chemcraft видно, что оптимизация прошла корректно, констрейны сработали, а молекула ни во что не изомеризовалась (ковалентные связи те же, что и на схеме выше), значит файлы XYZ и xtb-control получились корректными, и можно запускать конформационный поиск в программе CREST: <code>screst input.xyz xtb-control.txt</code>. Если в логе CREST присутствуют проблемы с топологией, то есть <code>screst_noreftopo</code>, он запустит crest в очередь с флагом <code>--noreftopo</code>; но с ним нужно быть аккуратным: часто проблемы с топологией возникают из-за того, что не прочитались констрейны из <code>xtb-control.txt</code>, в этом случае <code>--noreftopo</code> только навредит, замаскировав ошибку, и приведя к неправильным результатам в конформационном поиске. | ||
# В результате конформационного поиска (он может продолжаться несколько часов) CREST выдаст файл crest_conformers.xyz с найденными им конформациями. Эти конформации надо посмотреть в ChemCraft и если с ними всё в порядке - поставить поиски всех соответствуюущих переходных состояний (уже без констрейнов) в Orca, по аналогии с HomeWork1. Уровень теории PBE0-D3/def2SVP/PCM(H2O). В итоге вы получите все конформеры переходного состояния в рассматриваемой реакции и сможете определить, как же в нём всё таки развёрнуты OH-группы. | # В результате конформационного поиска (он может продолжаться несколько часов) CREST выдаст файл crest_conformers.xyz с найденными им конформациями. Эти конформации надо посмотреть в ChemCraft и если с ними всё в порядке - поставить поиски всех соответствуюущих переходных состояний (уже без констрейнов) в Orca, по аналогии с HomeWork1. Уровень теории PBE0-D3/def2SVP/PCM(H2O). В итоге вы получите все конформеры переходного состояния в рассматриваемой реакции и сможете определить, как же в нём всё таки развёрнуты OH-группы. | ||
В отчёте по этому ДЗ нужно подробно описать ход работы со скринами, определить самое низкоэнергетическое переходное состояние, рассчитать его вклад в протекание реакции, а также вклады всех остальных переходных состояний; определить, сколько переходных состояний нужно учесть, чтобы описать 99% протекания реакции. Отчёт присылайте на почту ccc@theorchem.ru. | В отчёте по этому ДЗ нужно подробно описать ход работы со скринами, определить самое низкоэнергетическое переходное состояние, рассчитать его вклад в протекание реакции, а также вклады всех остальных переходных состояний; определить, сколько переходных состояний нужно учесть, чтобы описать 99% протекания реакции. Отчёт присылайте на почту ccc@theorchem.ru. | ||
Revision as of 10:47, 9 October 2022
Задачей данного практического задания является нахождение наиболее стабильного переходного состояния (ПС) в сложной реакции SN2 замещения CN на Cl в модельном диоле:
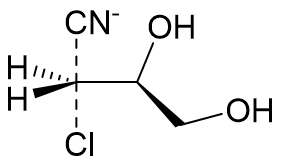
Очевидно, что в данном случае обе OH-группы могут образовывать водородные связи с первым или вторым нуклеофилом (они оба имеют частичный отрицательный заряд в ПС). Поэтому предсказать, как будет выглядеть переходное состояние, очень сложно. Но это можно установить, проведя конформационный поиск ПС и локализовав все конформеры ПС с помощью квантовохимических расчётов.
Для конформационного поиска ПС мы будем использовать разработанный Медведевым М.Г. и Пановой М.В. алгоритм, в котором изменяющиеся (разрывающиеся или замыкающиеся) связи зафиксированы (законстрейнены) на длинах, близких к реализующимся в переходных состояниях:
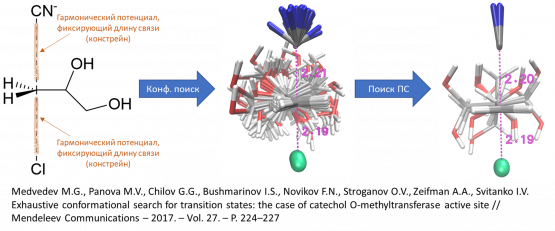
| Связь | Величина констрейна |
|---|---|
| H ... 2nd row atom | 1.4 Ангстрем |
| 2nd row atom ... 2nd row atom | 2.0 Ангстрем |
| 2nd row atom ... 3rd row atom | 2.3 Ангстрем |
Данный подход позволяет применять любые методы конформационного поиска для поиска ПС. В рамках данной практической работы конформационный поиск будет выполняться в программе CREST, а последующая оптимизация гессов (стартовых структур) - в Orca.
Процесс выполнения
- Для начала нужно нарисовать структуру в ChemCraft. В переходном состоянии все показанные палочками длины связей должны быть близки к равновесным значениям (их можно посмотреть вот тут), а разрывающиеся/замыкающиеся связи C...C и C...Cl имеют длины 2.0 и 2.3 ангстрем, соответственно. Также стоит обратить внимание на формы атомов (так, sp3-углероды должны быть тетраэдрическими) и торсионные углы: как правило, заторможенные конформации предпочтительнее заслонённых.
- Чтобы научиться им пользоваться, выполните все задачи из этого мануала.
- Следующая задача: проверить корректность построенной молекулы и выбранных параметров (force constant) перед конф. поиском путём запуска оптимизации в XTB. Запуск XTB требует двух файлов, XYZ и xtb-control.txt (они могут называться и иначе, но файл XYZ обязательно должен иметь именно такое расширение). Запуск XTB:
sxtb input.xyz xtb-control.txt
xtb-control.txt - это обычный текстовый файл, содержащий:Файлы XYZ очень широко используются для обмена информации в квантовой химии и имеют следующую структуру:$chrg -1 $constrain distance: <номер углерода CN>, <номер центрального углерода>, auto distance: <номер атома хлора>, <номер центрального углерода>, auto angle: <номер атома хлора>, <номер центрального углерода>, <номер углерода CN>, auto force constant=2.0 $end
В XYZ-файле в строке 1 идёт число атомов в первой молекуле, далее в строке 2 - комментарий, содержащий любые символы, кроме переноса строки, затем следуют строчки, содержащие название элементов и их координаты X, Y и Z. После завершения первой молекулы (считается из количества атомов в ней), идёт вторая молекула, и так далее - сколько угодно молекул в одном XYZ-файле.2 Oxygen molecule O 0.000000000 0.000000000 0.000000000 O 0.000000000 0.000000000 1.300000000 3 Acetylene molecule C 0.695770838 0.000000000 -1.221910046 C 0.695770838 0.000000000 -0.016562046 H 0.695770838 0.000000000 1.049156954 H 0.695770838 0.000000000 -2.287629046
- После запуска расчёта в XTB, ждём его окончания, переименовываем полученный xtbopt.log в xtbopt_trj.xyz, скачиваем себе на компьютер, открываем в ChemCraft и смотрим, как шла оптимизация.
- Если в визуализации в Chemcraft видно, что оптимизация прошла корректно, констрейны сработали, а молекула ни во что не изомеризовалась (ковалентные связи те же, что и на схеме выше), значит файлы XYZ и xtb-control получились корректными, и можно запускать конформационный поиск в программе CREST:
screst input.xyz xtb-control.txt. Если в логе CREST присутствуют проблемы с топологией, то естьscrest_noreftopo, он запустит crest в очередь с флагом--noreftopo; но с ним нужно быть аккуратным: часто проблемы с топологией возникают из-за того, что не прочитались констрейны изxtb-control.txt, в этом случае--noreftopoтолько навредит, замаскировав ошибку, и приведя к неправильным результатам в конформационном поиске. - В результате конформационного поиска (он может продолжаться несколько часов) CREST выдаст файл crest_conformers.xyz с найденными им конформациями. Эти конформации надо посмотреть в ChemCraft и если с ними всё в порядке - поставить поиски всех соответствуюущих переходных состояний (уже без констрейнов) в Orca, по аналогии с HomeWork1. Уровень теории PBE0-D3/def2SVP/PCM(H2O). В итоге вы получите все конформеры переходного состояния в рассматриваемой реакции и сможете определить, как же в нём всё таки развёрнуты OH-группы.
В отчёте по этому ДЗ нужно подробно описать ход работы со скринами, определить самое низкоэнергетическое переходное состояние, рассчитать его вклад в протекание реакции, а также вклады всех остальных переходных состояний; определить, сколько переходных состояний нужно учесть, чтобы описать 99% протекания реакции. Отчёт присылайте на почту ccc@theorchem.ru.