Open: Blender. Визуализация молекул и орбиталей
Плюсы и минусы визуализации своих результатов в Blender
Плюсы:
- Полный контроль над тем, какой будет конечный результат. Кастомизировать можно практически всё, что угодно, так как blender сам по себе - это программа 3d моделирования общего назначения, а модель молекулы создатся пользовательскими python-скриптами.
- Легкая автоматизация. Рендер каждого кадра анимации (точки траектории молдинамики, скана или IRC) можно легко автоматизировать. Вручную нужно будет только один раз выбрать положение камеры (угол, под которым смотрим на молекулу), материалы атомов, цвет или прозрачность фона.
Минусы:
- Пока не нашел.
Установка
На Windows
1. Скачайте установщик или portable-версию по ссылке.
2. Установите/распакуйте архив и добавьте путь к blender.exe в переменную Path (см. здесь).
3. Убедитесь, что блендер запускается из командной строки, если написать там "blender.exe". Должно появиться окно программы (Рис. 1).
На Linux
Установка на кластер и личный компьютер делается по одинаковому принципу:
1. Скачать архив с программой (см. список версий) и распаковать:
wget https://download.blender.org/release/Blender2.93/blender-2.93.4-linux-x64.tar.xz tar -xf blender-2.93.4-linux-x64.tar.xz
2. Добавить полный путь в полученную директорию blender-2.93.4-linux-x64 в PATH.
3. Проверить, что получается запустить блендер командой blender (в случае личного компьютера) и blender -b (в случае расчетного компьютера). Флаг -b отключает отображение графического интерфейса, что полезно, когда работаете по ssh-соединению. Если всё работает, результат должен быть примерно таким:
[user@server any_directory]$ blender -b Blender 3.3.1 (hash b292cfe5a936 built 2022-10-05 00:14:35) /run/user/1000/gvfs/ non-existent directory Blender quit [user@server any_directory]$
В случае запуска на персональном компьютере без флага -b должно появиться окно программы (Рис. 1).
Пререквизиты Python (нужно для всех задач и под любой ОС)
pip install numpy scipy networkx pandas pymcubes scikit-learn plotnine imageio
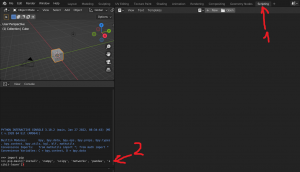
Так как в блендере есть свой встроенный Python, то в него нужно тоже установить эти пакеты (см. картинку):
import sys, subprocess
subprocess.call([sys.executable, '-m', 'ensurepip'])
subprocess.call([sys.executable, '-m', 'pip', 'install', 'numpy', 'scipy', 'networkx', 'pandas', 'scikit-learn', 'plotnine', 'imageio'])
После запуска команд subprocess.call надо немножко подождать - интерфейс зависает на время установки пакетов. Если работаете на кластере, то можно установить пакеты и не вызывая графический интерфейс - запустите Python-интерпретатор блендера /path/to/blender/python/bin/python3.9 и выполните все те же команды.
После установки попробуйте импортировать, чтобы убедиться, что всё установилось:
import numpy, scipy, networkx, pandas, sklearn, plotnine, imageio
Есть известная проблема, когда pip install пишет, что пакеты уже установлены, но импортировать их не удается. Дело в том, что блендер видит пакеты в вашем системном python, но не может импортировать. Это решается запуском блендера с дополнительным флагом --python-use-system-env.
Пререквизиты для дополнительных расчетов
rung
В некоторых местах этого мануала понадобится дополнительное выполнение квантовохимических расчетов в Гауссиане, для этого нужно добавить в PATH путь к скрипту с названием rung. Этот скрипт должен брать на вход название gjf-файла и запускать для него расчет. Содержание скрипта rung должно быть примерно таким (проверьте переменные GVER, GAUSS_EXEDIR и G09BASIS):
#!/bin/bash
INPFILE=$1
GVER=g16avx
export GAUSS_SCRDIR=/scratch/gaussian
export GAUSS_EXEDIR="/opt/"$GVER""
export GAUSS_ARCHDIR="/"$GVER"/arch"
export GMAIN=$GAUSS_EXEDIR
export LD_LIBRARY_PATH=$GAUSS_EXEDIR
export G09BASIS="/opt/"$GVER"/basis"
export F_ERROPT1="271,271,2,1,2,2,2,2"
export TRAP_FPE="OVERFL=ABORT;DIVZERO=ABORT;INT_OVERFL=ABORT"
export MP_STACK_OVERFLOW="OFF"
export KMP_DUPLICATE_LIB_OK="TRUE"
export PATH=${PATH}:${GAUSS_EXEXDIR}
/opt/"$GVER"/g16 $INPFILE
NBO6
Архив с NBO6 -- Nbo6_linux.zip (File:Nbo6 linux.zip)
NBO6 требуется для работы скрипта, автоматизирующего генерацию данных о МО. Для запуска расчетов нужно, чтобы в PATH был путь к скрипту NBO6 из архива Nbo6_linux.zip, а в скрипте NBO6 было правильное значение NBOBIN (директория с nbo6.exe) и актуальный путь к Intel MKL.
Чтобы проверить работоспособность NBO6, можете запустить расчет для файла test.47 из архива Nbo6_linux.zip:
NBO6 test.47
Рисуем молекулу
Архив с примерами и скриптами -- Blender_si.zip (File:Blender si.zip)
Скачайте репозиторий со вспомогательными скриптами:
git clone https://github.com/nkrivoshchapov/chemscripts.git
Важный комментарий по продолжению мануала: ниже вас ждет куча питоновских скриптов. Их идентичные копии лежат в архиве мануала, так что нет необходимости копипастить код с веб-страницы. Во-вторых, обратите внимание, по какому принципу будет оформлен код:
# ./путь/к/скрипту_в_архиве_мануала.py
import A as B # Импорты и, возможно, ещё какой-то код
from C import D, E, F
# Все переменные, которые названы капсом, задаются где-то в начале скрипта
# и их значения вам нужно указать самостоятельно
INPUT_FILENAME = "my_amazing_molecule.sdf"
OUTPUT_FILENAME = "new_scene.blend"
OTHER_USER_DEFINED_DATA = [1, 2, 3]
#
# А дальше уже идет сама программа
#
Создание MOL файла
Прежде чем рисовать молекулу, нужно каким-то образом сгенерировать связи между атомами, так как обычные XYZ-файлы и логи квантовохимических оптимизаций этой информации не содержат. Самый простой способ - это открыть структуру в программе типа ChemCraft или GaussView и сохранить файл типа .MOL или .SDF. Файлы с этим форматом содержат в дополнение к координатам информацию о связях, которые ChemCraft (или другая программа) сгенерировали на основании межатомных расстояний.
Способ через ChemCraft не всегда подходит, так как иногда файлов бывает слишком много. Ниже приведено два автоматических способа генерации связей из структуры молекулы.
Через межатомные расстояния
Это наиболее быстрый способ. Допустим, нужно сгенерировать MOL файл из лога оптимизации Гауссиана. Для этого нужно выполнить следующий код (замените STARTFILE на название лог-файла):
# ./molecule/generate_sdf.py
from chemscripts import utils, geom
STARTFILE = "./meoome.log" # The name of optimization log-file
if __name__ == "__main__":
xyzs, syms = utils.parse_gaussian_log(STARTFILE)
curmol = geom.Molecule(shutup=True)
curmol.from_xyz(xyzs, syms, mult=1.2)
sdfname = STARTFILE.replace('.log', '.sdf')
curmol.save_sdf(sdfname)
Через топологию из расчета NBO
Этот способ обычно дает более правильные связи, но требует выполнения дополнительного расчета SCF и NBO. Допустим, нужно сгенерировать MOL файл из лога оптимизации Гауссиана. Для этого выполните следующий код (замените STARTFILE на имя лог-файла):
# ./molecule/generate_sdf2.py
import sys
from chemscripts.calc import queue
from chemscripts.mylogging import createLogger
from chemscripts import utils, geom
STARTFILE = "./meoome.log" # The name of optimization log-file
if __name__ == "__main__":
for exe in ("rung",):
assert utils.check_availability(exe), "%s is not included in PATH" % exe
if len(sys.argv) > 1:
maxproc = int(sys.argv[1])
else:
maxproc = None
gdriver = queue.init_thread(maxproc)
logger = createLogger("Main")
logger.info("Step 1. Run NBO3 analysis for initial geometry")
logger.info("Processing " + STARTFILE)
xyzs, syms = utils.parse_gaussian_log(STARTFILE)
gjfname = STARTFILE.replace('.log', '_nbo3.gjf')
utils.write_gjf(xyzs, syms, 'nbosimple', gjfname,
subs={
'nproc': 1,
})
nbogjfs = [gjfname]
utils.start_calcs(nbogjfs, gdriver)
logger.info("Step 1 is finished")
logname = gjfname.replace('.gjf', '.log')
logger.info("Step 2. Generate SDFs with topologies from NBO for Blender")
logger.info("Reading " + logname)
curmol = geom.Molecule(nbolog=logname)
curmol.from_xyz(xyzs, syms)
sdfname = STARTFILE.replace('.log', '.sdf')
curmol.save_sdf(sdfname)
logger.info("Step 2 is finished")
logger.info("My work is done. Terminating.")
with gdriver['todo_lock']:
gdriver['todo_files'].append("Done")
Генерируем сцену
Для начала понадобится файл scene_template.blend, который лежит в архиве мануала. Там есть пустая сцена, в которой только камера и источник освещения, а также шаблоны материалов орбиталей, которые понадобятся в следующем разделе. После того, как раздобыли scene_template.blend нужно такой скрипт и установить в нём желаемые значения для SDFNAME, RESFILE и SMALL_ATOMS:
# ./molecule/draw_molecule.py
import bpy
from importlib import reload
import sys, os
mycwd = os.getcwd()
if mycwd not in sys.path:
sys.path.insert(0, mycwd)
import chemscripts.blender as msblend
msblend.cleanup()
SDFNAME = "./meoome.sdf" # The file generated earlier
SMALL_ATOMS = [] # Atoms that have to be smaller than others, indices start from 1
RESFILE = "meoome_scene.blend" # Filename for the generated scene
m = msblend.Molecule(small_atoms=SMALL_ATOMS)
m.read_sdf(SDFNAME)
m.draw_bonds(caps=False, radius=0.1)
m.draw_atoms(scale=0.35)
bpy.ops.wm.save_as_mainfile(filepath=os.path.join(os.getcwd(), RESFILE))
Выполните скрипт следующей командой:
blender scene_template.blend -b -P draw_molecule.py
Если всё прошло успешно, то должен появится файл с названием, заданным в RESFILE, и в терминале появится примерно это:
Blender 2.93.4 (hash b7205031cec4 built 2021-08-31 23:43:17) Read prefs: /home/username/.config/blender/2.93/config/userpref.blend Read blend: /.../molecule/scene_template.blend Warning: No mesh data to join Warning: No mesh data to join 0.19109725952148438 seconds Info: Total files 0 | Changed 0 | Failed 0 Info: Saved "meoome_scene.blend" Info: Saved "meoome_scene.blend" Blender quit
Объясню подробнее, что сейчас произошло. Сначала блендер прочитал флаг -b, который означает, что мы не хотим, чтобы открывался графический интерфейс. Потом блендер открыл сцену из файла scene_template.blend, после чего он начал выполнять скрипт из файла draw_molecule.py. Код из этого скрипта сделал модель молекулы из файла SDFNAME и сохранил измененную сцену в RESFILE.
Рендер на локальном компьютере
Откройте полученный файл в блендере, например, командой:
blender meoome_scene.blend
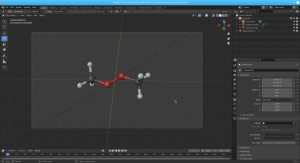
Когда появится окно блендера, вы увидите модель вашей молекулы. Очень рекомендую вам разобраться в способах навигации по сцене блендера - это сейчас пригодится (см. видеоуроки).

Чтобы получить картинку молекулы выполните следующее
1. Найдите хороший угол обзора молекулы. Его можно менять, если зажать колесико и двигать мышью.
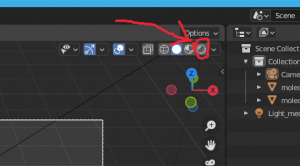
2. Выделите камеру и нажмите Ctrl+Alt+NumPad0 (клавиша ноль на numpad'e), чтобы поставить камеру на место, с которого вы сейчас смотрите на молекулу (результат - Рисунок 2). Ctrl+NumPad0 - переход к обзору текущего положения камеры.
3. Теперь измените тип Viewport shading с Solid на Rendered (Рисунок 3). Картинка превратится в немного шумный вариант того, какой она будет после рендера. Если вас всё устраивает, жмите F12, а потом - Save As (Рисунок 4).
Рендер на удаленном компьютере
Выполните на своем компьютере 1 и 2 шаги из предыдущего подраздела. Таким образом, вы подготовите положение камеры и останется только выполнить рендер. Нажмите Ctrl+S и загрузите файл со сценой на удаленный компьютер. Затем создайте такой скрипт:
# ./molecule/just_render.py
import bpy, os
NTHREADS = 8 # Number of threads
PNGNAME = "meoome_rendered.png" # Name of the final image
if __name__ == "__main__":
render = bpy.context.scene.render
render.threads = NTHREADS
render.threads_mode = 'FIXED'
render.image_settings.file_format = 'PNG'
render.filepath = os.path.join(os.getcwd(), PNGNAME)
bpy.ops.render.render(write_still=1)
И выполните его на удаленном компьютере в сцене, которую вы создали на своём локальном:
blender meoome_scene.blend -b -P just_render.py
В итоге получится картинка с названием, указанным в PNGNAME.
Рисуем молекулу с орбиталями
Как и в случае молекулы без орбиталей, мы сначала генерируем входные данные для создания 3D модели. Однако теперь нам нужны не только данные о связях в молекуле, а ещё и 3D данные о поверхности орбиталей (вершины, ребра и грани), для генерации которых нужно будет провести несколько дополнительных расчетов.
Генерация 3D данных об орбиталях. NBO.
Создайте и выполните следующий скрипт. Обратите внимание, что требуется присутствие в PATH трех программ/скриптов: rung, formchk, cubegen. Про первый скрипт говорилось выше, а вторые два лежат в папке с Гауссианом.
# ./orbitals/nbo.py
import os, sys, copy, glob, ntpath
import shutil, json
from chemscripts.calc import queue
from chemscripts.mylogging import createLogger
from chemscripts.nbo import NBO3LogParser, highest_p_character, generate_nbo_cube, generate_isosurface
from chemscripts import utils, geom
STARTFILE = "./meoome.log" # The name of optimization log-file
CALCDIR = "./calc_files" # Dir for all new files
NBO_MASKS = {
'donor': r"LP\(.*\)O5$", # Regex for the NBO of interest
'acceptor': r"BD\*\(1\)C7-H8$", # Another one
}
ISOVALUE = 0.15 # Isovalue for all NBOs
ASSIGNMENT_FNAME = "./nbo_assignment.json"
if __name__ == "__main__":
# Длинный скрипт. Не буду приводить целиком, но шаги следующие:
# 1. Run NBO3 analysis for initial geometry
# 2. Obtain formatted Gaussian checkpoints
# 3. Obtain indices of NBOs of interest
# 4. Generate cube-files for chosen NBOs
# 5. Generate isosurface data from cube-files
# 6. Generate SDF based on topology from NBO
После запуска скрипта будет создана директория CALCDIR, в которой будут лежать нужные нам файлы (ненужные не приведены):
meoome_nbo3_15_plus.*iso - три файла с данными о вершинах, ребрах и гранях поверхности 15ой NBO (isovalue = ISOVALUE); meoome_nbo3_15_minus.*iso - аналогично, но для уровня противоположного знака (isovalue = -ISOVALUE); meoome_nbo3_15_*.*iso - аналогично, но для 128ой NBO; meoome.sdf - файл с геометрией, из которого будут взяты координаты и связи.
Номера и количество орбиталей будут отличаться в зависимости от молекулы и от кого, как вы модифицируете скрипт под свою задачу - сейчас nbo.py расчитан на рисование двух орбиталей, основываясь на регулярных выражениях для их обозначений в логе (см. код).
Помимо этого, будет сгенерирован файл ASSIGNMENT_FNAME, необходимый для сопоставления названий орбиталей ('donor', 'acceptor' и т.п.) их численным индексам, которые используются в названиях файлов изоповерхностей.
Генерация 3D данных об орбиталях. MO.
Для запуска скрипта генерации МО требуется присутствие в PATH четырех программ/скриптов: rung, formchk, cubegen, NBO6. Про первый скрипт говорилось выше, а вторые два лежат в папке с Гауссианом. NBO6 - это скрипт, который запускает NBO6 и пишет лог в файл *_NBO.out, у него должно быть примерно следующее содержание:
#!/bin/bash
export NBOBIN=/opt/NBO6
# path of Intel MKL
export LD_LIBRARY_PATH=/opt/intel/compilers_and_libraries_2019.5.281/linux/mkl/lib/intel64_lin:$LD_LIBRARY_PATH
export NBOEXE=$NBOBIN/nbo6.exe
export GENEXE=$NBOBIN/gennbo6.exe
export PATH=$PATH:$NBOBIN
$GENEXE $1 > ${1%.*}_NBO.ou
Скрипт для генерации МО:
# ./orbitals/mo.py
# A lot of imports...
# User-defined data:
STARTFILE = "./meoome.log" # The name of optimization log-file
CALCDIR = "./calc_files" # Dir for all new files
MO_IDXS = ["LUMO+1", "LUMO", "HOMO", "HOMO-1", "HOMO-2", "HOMO-3"] # Desired MOs
ISOVALUE = 0.08 # Isovalue for all MOs
ASSIGNMENT_FNAME = "./mo_assignment.json"
# Очень большой скрипт, но суть такая:
# 1. Запустить single-point расчет, чтобы было откуда брать МО
# 2. Запустить расчет NBO3, чтобы получить входные данные для NBO6
# 3. Запустить расчет NBO6, чтобы взять оттуда занятости и энергии МО
# 4. Сконвертировать chk->fchk, чтобы было для чего запускать cubegen
# 5. Понять для каких индексов МО нужно генерировать поверхности (записать сопоставление название <-> индекс)
# 6. Запустить cubegen для МО с выбранными индексами
# 7. Сгенерировать поверхности с заданным ISOVALUE для всех cube-файлов
# 8. Сгенерировать SDF, взяв топологию из уже имеющихся логов NBO3.
Генерируем сцену
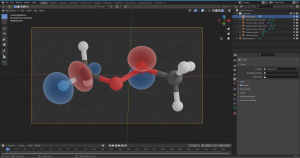
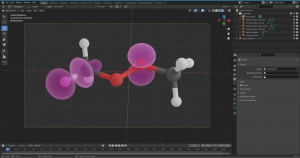
Как и в случае с молекулой без орбиталей, на следующем шаге нужно подготовить скрипт для генерации вашей 3D модели: проверьте входные данные (немного отличаются для NBO и MO), а также выберите стиль рисования орбиталей в конце скрипта.
# ./orbitals/draw_orbitals.py
import bpy
import sys, os, json, ntpath
mycwd = os.getcwd()
if mycwd not in sys.path:
sys.path.insert(0, mycwd)
import chemscripts.blender as msblend
import chemscripts.blender.draw_objects.orbitals as msorbital
msblend.cleanup()
SDFNAME = "./calc_files/meoome.sdf" # The file generated earlier
SMALL_ATOMS = [] # Atoms that have to be smaller than others, indices start from 1
RESFILE = "meoome_scene.blend" # Filename for the generated scene
ASSIGNMENT_FNAME = "./nbo_assignment.json" # or "./mo_assignment.json"
CALCDIR = "./calc_files" # Dir for all new files
PLOT_ORBITALS = ["donor", "acceptor"] # Names of NBOs
# PLOT_ORBITALS = ["HOMO", "LUMO"] # or names of MOs
with open(ASSIGNMENT_FNAME, 'r') as f:
orbital_data = json.load(f)
molname = ntpath.basename(SDFNAME).replace('.sdf', '')
m = msblend.Molecule(small_atoms=SMALL_ATOMS)
m.read_sdf(SDFNAME)
m.draw_bonds(caps=False, radius=0.1)
m.draw_atoms(scale=0.35)
for orbname in PLOT_ORBITALS:
orbidx = orbital_data[molname][orbname]
if "HOMO" in orbname or "LUMO" in orbname:
filenames = f"{molname}_scf_{orbidx}" # For MOs
else:
filenames = f"{molname}_nbo3_{orbidx}" # For NBOs
# 1) Different colors for different signs
# msorbital.plot_nbo(filenames, nbodir=CALCDIR, color=["#DF4A4A", "#64A7E7"], reverse=False)
# 2) Monochromatic style
msorbital.plot_nbo(filenames, nbodir=CALCDIR, color="#f363e0", reverse=False)
# 3) To get zebra-like orbitals: color=["#111111", "#FFFFFF"], wavy=True
bpy.ops.wm.save_as_mainfile(filepath=os.path.join(os.getcwd(), RESFILE))
Команда для генерации сцены аналогично тому, что уже делали выше:
blender scene_template.blend -b -P draw_orbitals.py
Скорее всего, вам хотелось бы рисовать разные орбитали разными цветами, но, надеюсь, вы справитесь сделать dict, в котором ключи - элементы PLOT_ORBITALS, а значения - то, что хотите передавать аттрибутом color=...
Все следующие действия полностью аналогичны рисованию без орбиталей.
Постобработка
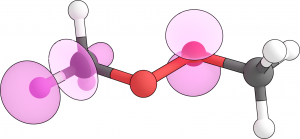
Может захотеться обрезать неинформативную часть получаемых картинок или сделать их более яркими. Для этого можно использовать следующий скрипт:
# ./postprocessing/image2image.py
import glob, os
from PIL import Image, ImageEnhance
from chemscripts import imageutils
IMAGE_MASK = "./*_rendered.png" # Files to process
TO_FINAL = lambda old_name: old_name.replace("_rendered.png", "_final.png") # Rule for renaming
BRIGHTNESS = 1.3
if __name__ == '__main__':
for pngname in glob.glob(IMAGE_MASK):
main_image = Image.open(pngname)
main_image.load()
# Fills the transparency
bg = Image.new("RGB", main_image.size, (255, 255, 255))
bg.paste(main_image, mask=main_image.split()[3])
main_image = bg
# Increases brightness
enhancer = ImageEnhance.Brightness(main_image)
main_image = enhancer.enhance(BRIGHTNESS)
# Trims white space
trimbox = imageutils.TrimmingBox()
trimbox.extend(main_image)
main_image = main_image.crop(trimbox.points)
newpngname = TO_FINAL(pngname)
main_image.save(newpngname, 'PNG', quality=100)
Дополнения
Здесь лежат нетестированные идеи, что можно ещё делать в блендере.
Рендер на видеокарте
Блендер позволяет рендерить на видеокарте и это сильно быстрее.
1. В настройках включить видеокарту:
- Edit-Preferences-System-Opix (Nvidia GPU)
- Edit-Preferences-System-HIP (AMD GPU)
- Edit-Preferences-System-oneAPI (Intel GPU)
2. Скопировать файл userpref.blend на сервер (см. здесь)
3. Включить видеокарту в настройках рендера и сохранить такой blend файл
TODO
1. Анимации
2. Кратные связи
3. GPU
4. New style and switchable transparency